Un estudio de investigación colaborativo entre equipos de investigación de la Pontificia Universidad Católica (PUC), la Universidad Andrés Bello (UNAB) y el Centro INCAR, permitió desarrollar un nuevo protocolo de PCR multiplex para tipificar los sub-linajes existentes en Chile del agente del BKD, Renibacterium salmoninarum.
La investigación que tiene por autores a Patricia Mora-Salas, Sebastián Zapararte, Pamela Villouta, Henry Araya-León, Ruben Avendaño-Herrera, Francisco Melo, Fernando O. Mardones, quienes crearon un novel protocolo de PCR multiplex in silico y lo validaron a partir del trabajo de laboratorio con 27 aislados de R. salmoninarum chilenos recuperados de muestra de riñón, corazón, hígado y/o bazo de salmón Atlántico y salmón Coho cultivados desde la Región de La Araucanía hasta la Región de Magallanes durante brotes ocurridos entre 2014 y 2021.
Los expertos calificaron este enfoque de secuenciación multiplex PCR-Sanger “como una herramienta valiosa para la tipificación rápida y confiable de cada uno de los cuatro sub-linajes descritos para aislados chilenos de R. salmoninarum”. Asimismo, los investigadores destacaron que “el método de tipificación de sub-linaje tiene un costo menor y requiere menos trabajo y esfuerzo en comparación con la tarea de secuenciar el genoma completo de una muestra y facilitará futuros estudios epidemiológicos retrospectivos o prospectivos que involucren infecciones por R. salmoninarum en pisciculturas chilenas y centros de engorda”.
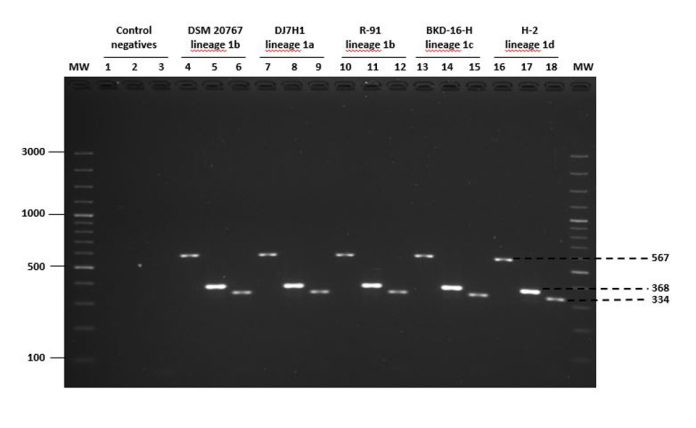
Aunque el genoma completo de R. salmoninarum (~3,1 millones de nucleótidos) está altamente conservado, estudios previos de epidemiología genómica identificó dos linajes (1 y 2) y dentro del linaje 1 cuatro sub-linajes dentro de los que se encuentran los aislamientos chilenos: 1a, 1b, 1c y 1d. En el estudio, los investigadores de la PUC, UNAB e INCAR identificaron algunas secuencias de nucleótidos que presentaban diferencias en regiones bien específicas del genoma, comúnmente llamado polimorfismo de nucleótido único (en inglés SNPs).
Así, los científicos descubrieron un conjunto de tres nucleótidos (SNP 2088, 2092 y 2123) posicionados dentro del genoma en una forma que permitía su amplificación simultánea mediante PCR, y que a su vez eran únicos para cada sub-linaje permitiendo su diferenciación.
Dentro de las aplicaciones prácticas de estos resultados, el Investigador Principal del Centro INCAR y académico de la UNAB, Dr. Ruben Avendaño, explicó que conocer el sub-linaje de R. salmoninarum endémico de una piscicultura, permitiría realizar el seguimiento en caso de ocurrir brotes después del traslado de los salmones a engorda en mar, e incluso determinar si los salmones infectados pudieran tener bacterias de dos o más sub-linajes en un mismo centro o brote.
“El protocolo está validado y las aplicaciones son muy valiosas. Por ejemplo, los laboratorios de diagnóstico de la red podrían implementarlo para la caracterización de los linajes, asociándolos a las distintas pisciculturas o centros de engorda. Incluso un aislado que nosotros tenemos categorizado como altamente virulento presenta un linaje específico, por tanto, realizaremos más estudios nuevamente en colaboración para asociar el potencial patogénico de R. salmoninarum y sus sub-linajes”, explicó el Dr. Avendaño-Herrera.
Renibacterium salmoninarum es el agente etiológico de la enfermedad renal bacteriana (BKD), que afecta significativamente a los salmónidos de cultivo y silvestres en todo el mundo. En Chile, el impacto productivo del BKD no está cuantificado, pero las pérdidas por mortalidad en agua dulce se ven reflejadas en el crecimiento, afectando a un estimado de hasta un 20% de la población aproximadamente, dependiendo de la especie.
Revisa el paper “Method for lineage typing of epidemic Renibacterium salmoninarum in Chilean salmon farms”, aquí:
https://onlinelibrary.wiley.com/doi/full/10.1111/jfd.13761